
2023年12月8日
国立研究開発法人 国立国際医療研究センター(NCGM)
国立研究開発法人 国立循環器病研究センター(NCVC)
国立研究開発法人 国立精神・神経医療研究センター(NCNP)
国立研究開発法人 国立成育医療研究センター(NCCHD)
国立研究開発法人 国立長寿医療研究センター(NCGG)
印刷用PDF(1MB)
ナショナルセンター・バイオバンクネットワーク(NCBN)
の試料を使った全ゲノムシークエンス解析
の試料を使った全ゲノムシークエンス解析
研究成果のポイント
- 国立研究開発法人 国立国際医療研究センター(略称:NCGM)、国立研究開発法人 国立循環器病研究センター(略称:NCVC)、国立研究開発法人 国立精神・神経医療研究センター(略称:NCNP)、国立研究開発法人 国立成育医療研究センター(略称:NCCHD)、国立研究開発法人 国立長寿医療研究センター(略称:NCGG)は、ナショナルセンター・バイオバンクネットワークのバイオバンクの試料の全ゲノムシークエンス解析(WGS解析)*1を実施しました。WGS解析を5つのナショナルセンターが同一のプロトコル(手法)で実施することにより、均質なゲノム情報を取得できました。
- 今回のWGS解析で得られたゲノム情報はがんや難病/希少遺伝性疾患の研究のために必須なコントロール(対照)データとなります。
- 一方、今回のWGS解析で得られたゲノム情報は集団遺伝学*2の貴重な研究対象でもあるため、ゲノム情報を解析して日本人集団の遺伝的な特徴を研究しました。
- その成果の一つとして、ゲノム情報から過去の人口の変化を推定し、沖縄と沖縄以外の地域(本土)では人口変化のパターンが異なることを明らかにしました。
- もう一つの成果として、東アジア人において正の自然淘汰を受けていると考えられるアルコール代謝、メラニン代謝、脂肪酸代謝の遺伝子の過去の遺伝子頻度を推定し、2万5000年から1万年前の異なる時期に遺伝子頻度の上昇が始まったことを示しました。
研究の背景
近年、一人の人間のすべての塩基配列を読み取る全ゲノムシークエンス解析(WGS解析)が発展し、解析で得たゲノム情報から病気の原因を突き止め、その治療法を開発しようという研究がさかんになっています。我が国では2019年に「全ゲノム解析等実行計画」が策定され、がんと難病を対象としたプロジェクトが進行中です。
ただし、ゲノムに潜むがんや難病の原因を探るには、がんや難病にかかっている人と、そうではない人のゲノム情報を比べる必要があります。そこで、ナショナルセンター・バイオバンクネットワーク(略称:NCBN)は国の計画に協力し、「がんや難病ではない人」(コントロール群)のゲノム情報を取得しました。この解析で得られたゲノム情報はがんと難病のゲノム研究のコントロール(対照)データとして利用される予定です。
その一方で、多数の日本人のゲノム情報は、集団遺伝学の研究対象としても貴重なものです。特に、NCBNの6つのNCは東京都、愛知県、大阪府にあることから、WGS解析の対象者の出身地は全国に広がっており、そのゲノム情報は日本国内の遺伝的多様性を解析するのに適していると期待されます。
そこで今回の研究は、①WGS解析で得られたゲノム情報の品質を確認すること、②ゲノム情報の詳細な解析を行い、日本人の集団遺伝学的な特徴を明らかにすることを目的としました。
研究内容
NCBNは全国に6か所ある国立高度専門医療研究センター(ナショナルセンター、NC)が参加するネットワーク型・連邦型のバイオバンクの運営組織です。NCBNのバイオバンクにはこれまで137,236人(11月24日現在)の参加者から提供された試料や情報が保管されています。これらのうち、がんや難病の病歴のない9,850人のDNA試料を対象としてWGS解析を行いました(表)。
| バイオバンク | WGS解析数 |
| 国立循環器病研究センター(NCVC) | 2043 |
| 国立精神・神経医療研究センター(NCNP) | 1758 |
| 国立国際医療研究センター(NCGM) | 2044 |
| 国立成育医療研究センター(NCCHD) | 2115 |
| 国立長寿医療研究センター(NCGG) | 1890 |
| 合計 | 9850 |
表: 本研究で実施したWGS解析数と提供元バイオバンク
この解析は、NCBNのバイオバンクが連携して行った初めてのゲノム解析であり、5つのNCのバイオバンクがそれぞれの保有試料を解析する際に、同一の解析プロトコルを用いました。こうして得たゲノム情報について、以下の研究を行いました。
- WGS解析で得られたゲノム情報の品質チェックを行いました。様々な手法でチェックした結果、どの手法でもエラー率はかなり低いことがわかりました。また、5つのバイオバンク間でのばらつきは少ないことが明らかになり、同一のプロトコルを使用したことが奏功したと考えられました。さらに、NCBNのDNA試料には血液由来のものと唾液由来のものがあるので、その違いによる影響がないかを検討しました。唾液には口腔内の雑菌など様々な由来のDNAの影響が認められましたが、以後の解析への影響はほとんどありませんでした。
- 個人間の塩基配列の違いを調べました。ヒトゲノムは30億塩基からなっていますが、標準的な配列と比べると、塩基の種類が異なっていたり、抜けていたり、余分に入っていたりする箇所が誰でもあります(これを「バリアント」と呼びます)。本研究で得たゲノム情報を解析したところ、合計で約1億573万か所のバリアントが見つかりました。そのうち45.8%は公的データベースに登録のない新規のバリアントで、そのほとんど(99.99%)は頻度が低く(集団中のアリル頻度が0.5%以下)、67.5%はただ一人にしか見つからない非常に稀なバリアントでした。このように稀なバリアントが見つかったのは、WGS解析の精度が高かったためと考えられます。
- 本研究で解析した対象者集団の遺伝学的な特徴を明らかにするために、国際1000人ゲノム計画*3の2,504人のデータと統合して集団遺伝学的解析を行いました。主成分分析(変数を減らすことでデータ分布の特徴を抽出しやすくする統計解析方法)によって、一人を一つの点としてプロットし、どのような集団構造が見られるかを解析しました。その結果、NCBNの集団は独自のクラスター(構造)を形成しており、国際1000人ゲノムのうちの東アジアの大陸集団(漢民族、ベトナムのキン族、シーサンパンナのダイ族)とは遺伝的に分化していることを再確認しました(図1)。
- ゲノム情報から過去の人口が世代ごとにどう変化してきたかを推定する方法を使って、主成分分析でクラスターを形成した沖縄と本土集団の過去の人口動態を推定しました。沖縄集団においては100世代前から25世代前に人口が減少した時期があり、本土集団では75世代から50世代前と17世代から11世代前の2回の人口減少期があることがわかりました(図2)。沖縄集団における人口減少と本土集団の1回目の人口減少は、ユーラシア大陸から日本列島への移動や九州から琉球列島への移動に伴う人口ボトルネック*4と考えられます。本土集団の2回目の人口減少は江戸時代に相当する期間に起こっています。この時期は人口増加の停滞が起こっていたことが知られており、歴史学的出来事がゲノムデータによって裏付けられた結果といえます。
- 過去の研究において、自然淘汰の影響を受けた東アジア人に固有な遺伝子が報告されています。今回の研究では、それらのうち、アルコール代謝に関連する遺伝子(ALDH2, ADH1B)、メラニン合成に関連する遺伝子(OCA2)、脂肪酸代謝に関連する遺伝子(FADS)に着目し、これらの遺伝子の集団内での遺伝子頻度が約3万年前から現在までにどう変化したかを推定しました。これらの遺伝子は2万5000年から1万年前に集団内で遺伝子頻度が急激に増えており、正の自然淘汰を受けていることを支持する結果でした。特にアルコール代謝に関する2つの遺伝子のうち、ADH1BのSNP(rs1229984) *5は約2万年前に頻度が上昇し始めたのに対して、ALDH2のSNP(rs671)は約7500年前とADH1Bより遅い時期に頻度上昇が始まっていました。このことは東アジア人の祖先集団においてアルコール代謝が関係する適応進化が段階的に起こったことを示しています。
今回の研究では、これらに加えてレアバリアントの影響の評価やヒト白血球型抗原遺伝子(HLA)の解析も行っており、論文で結果を報告しています
今回の研究では、これらに加えてレアバリアントの影響の評価やヒト白血球型抗原遺伝子(HLA)の解析も行っており、論文で結果を報告しています。
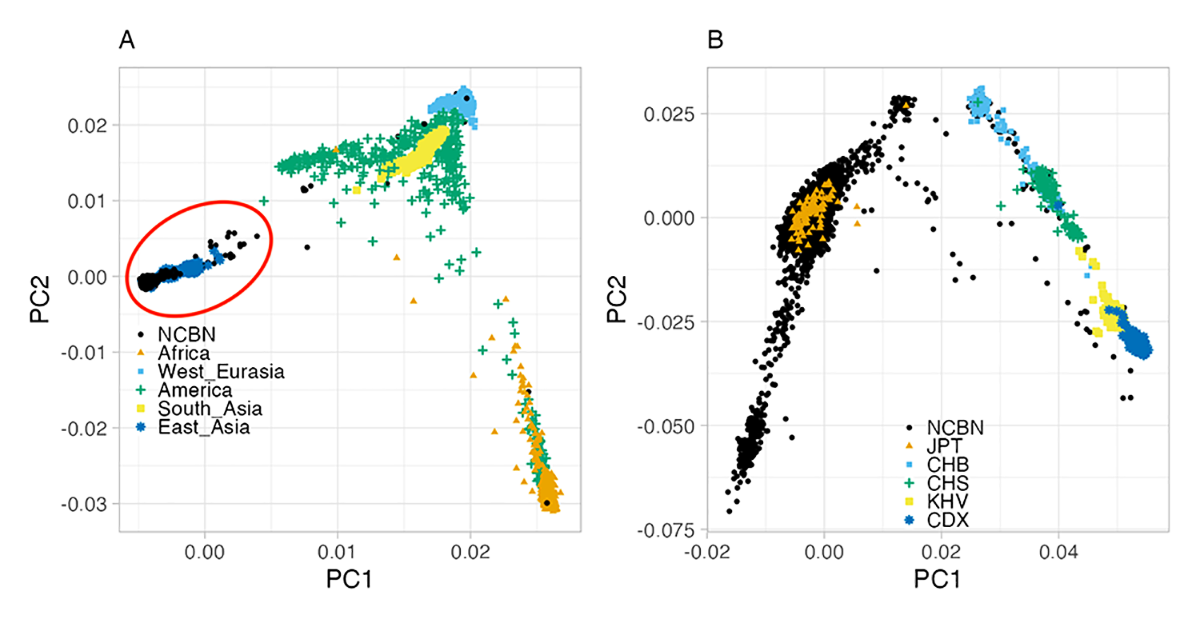
図1 主成分分析による集団構造解析
A:NCBNの解析対象者(黒)のクラスターと、国際1000人ゲノムのうちの東アジア集団(青)のクラスターは近いが、国際1000人ゲノムのうちのアフリカ、西ユーラシアなどの集団とは離れている。
B:Aのうち、赤丸で囲んだ部分をさらに主成分分析で分析したもの。NCBNの解析対象者(黒)と国際1000人ゲノムのうちの日本人(JPT、オレンジ)がクラスターをなしている。東アジア集団は別のクラスターを形成しているが、そのクラスターがさらに、中国北部の漢民族(CHB、ブルー)、中国南部の漢民族(CHS、緑十字)、ベトナムのキン族(KHV、黄色)、シーサンパンナのダイ族(CDX、青)のクラスターに分かれている。このような集団構造は、先行研究でも確認されていた。
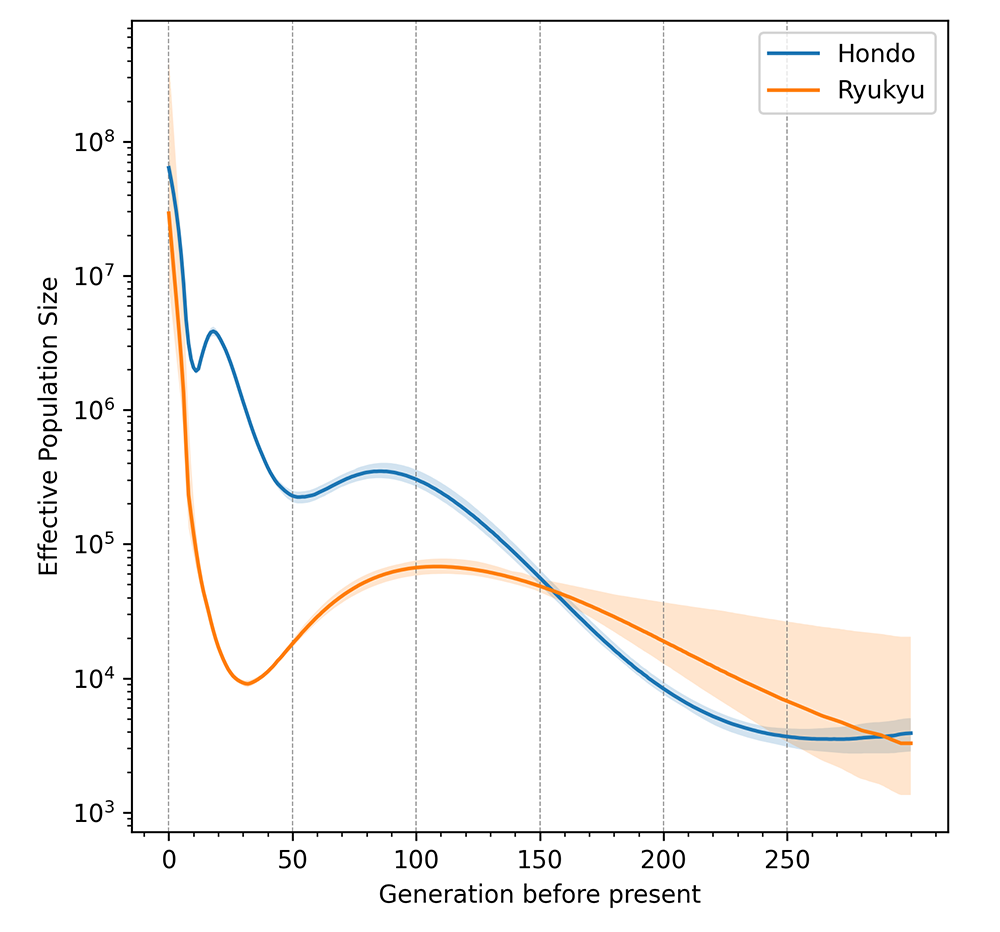
図2 ゲノム情報から推定した沖縄と本土の過去の人口変化
縦軸は人口の指標。横軸は世代数。左が現在で、右が過去。沖縄集団(オレンジ)では、100世代前から25世代前に人口減少期があり、本土集団(ブルー)では75世代から50世代前と17世代から11世代前の2回の人口減少期があることがわかる。
今後の期待
本研究ではNCBNの5つのバイオバンクの試料の全ゲノム解析を行いました。ナショナルセンターのバイオバンクには日本全国の様々な地域の出身者の試料が保管されており、今回の研究によって日本人集団の地域的多様性も明らかになりました。過去の人口変動の推定では沖縄と本土の集団の間で違いがあることを示しました。本研究で得られた全ゲノムデータはがんや難病などの疾患研究のコントロールとして活用される予定です。日本の地域的遺伝的多様性を含むNCBNのゲノムデータは地域的多様性を考慮した解析にも役立てられると期待されます。
発表雑誌
雑誌名:PLOS Genetics
論文タイトル:Exploring the genetic diversity of the Japanese Population: Insights from a Large-Scale Whole Genome Sequencing Analysis
掲載日:日本時間12月8日(金)午前4:00(米国東部時間12月7日午後2:00)オンライン版に掲載。
DOI:10.1371/journal.pgen.1010625
URL:https://journals.plos.org/plosgenetics/article?id=10.1371/journal.pgen.1010625
研究助成
本研究は日本医療研究開発機構・臨床ゲノム情報統合データベース整備事業(JP19kk0205012),文部科学省・新学術領域研究(18H05505)の支援を受けて行われました。
用語解説
*1 全ゲノムシークエンス解析(WGS解析):ゲノムの全長の塩基配列を次世代シークエンサーで解析する手法。
*2 ある集団の遺伝情報を統計的に解析することで、その集団の遺伝的な特徴の時間的変化や、他の集団との違いを明らかにする学問分野。
*3 国際1000人ゲノム計画: 世界の主要な集団を対象とした全ゲノム解析を行う国際共同研究。International 1000 Genomes Project.
*4 人口ボトルネック: 人口が減少してその後増加する現象。
*5 SNP:一塩基多型。スニップと読む。DNAの塩基配列における一塩基の違いのこと。rs1229984などは各SNPに固有の番号。
お問い合わせ先
(研究に関すること)
国立国際医療研究センター研究所 ゲノム医科学プロジェクト
(氏名)河合洋介
電話:03-5273-6858
E-mail: ykawai(a)ri.ncgm.go.jp
(取材に関すること)
国立研究開発法人 国立国際医療研究センター 企画戦略局広報企画室
電話:03-3202-7181
E-mail:press(a)hosp.ncgm.go.jp
国立研究開発法人 国立循環器病研究センター 企画経営部広報企画室
E-mail:kouhou(a)ml.ncvc.go.jp
国立研究開発法人 国立精神・神経医療研究センター 総務課広報室
E-mail:ncnp-kouhou(a)ncnp.go.jp
国立研究開発法人 国立成育医療研究センター 企画戦略局広報企画室
E-mail:koho(a)ncchd.go.jp
国立研究開発法人 国立長寿医療研究センター 総務部総務課総務係長(広報担当)
E-mail:webadmin(a)ncgg.go.jp
※E-mailは下記アドレス(a)の部分を@に変えてご使用ください。






